次世代シーケンサーとは
DNAの塩基配列を解読する装置のことをシーケンサーといいます。
なかでも次世代シーケンサーは、DNAの塩基配列を短時間で大量に解読できるため、2006年に登場して以降、生命科学や医学において広く使われています。
この記事では、次世代シーケンサーの原理や使用例、使用手順について解説します。
DNAの塩基配列を調べるシーケンサー
まず、シーケンサーについて解説します。シーケンサー(sequencer)とは、DNAの塩基配列を読み解く装置のことです。DNAの塩基には、アデニン(A)、チミン(T)、グアニン(G)、シトシン(C)という4種類があり、1列に並んで塩基配列を形成しています。この塩基配列を、「順序」「ひと続きのもの」という意味の英語であるシーケンス(sequence)とみなして、DNAの塩基配列を読み解くことをシーケンシング、読み解く装置のことをシーケンサーと呼びます。
DNAには遺伝子としての情報がコード(暗号化して保存)されており、DNAの塩基配列を知ることは遺伝情報を知ること、ひいては生命現象を知るための手掛かりとなります。
シーケンサーの歴史
1970年代に塩基配列を調べる方法がいくつか開発され、そのうち最も広まって現在でも使われている方法がジデオキシ法、またはこの技術を開発したイギリスの科学者フレデリック・サンガー(1918〜2013)の名を冠してサンガー法とよばれています。
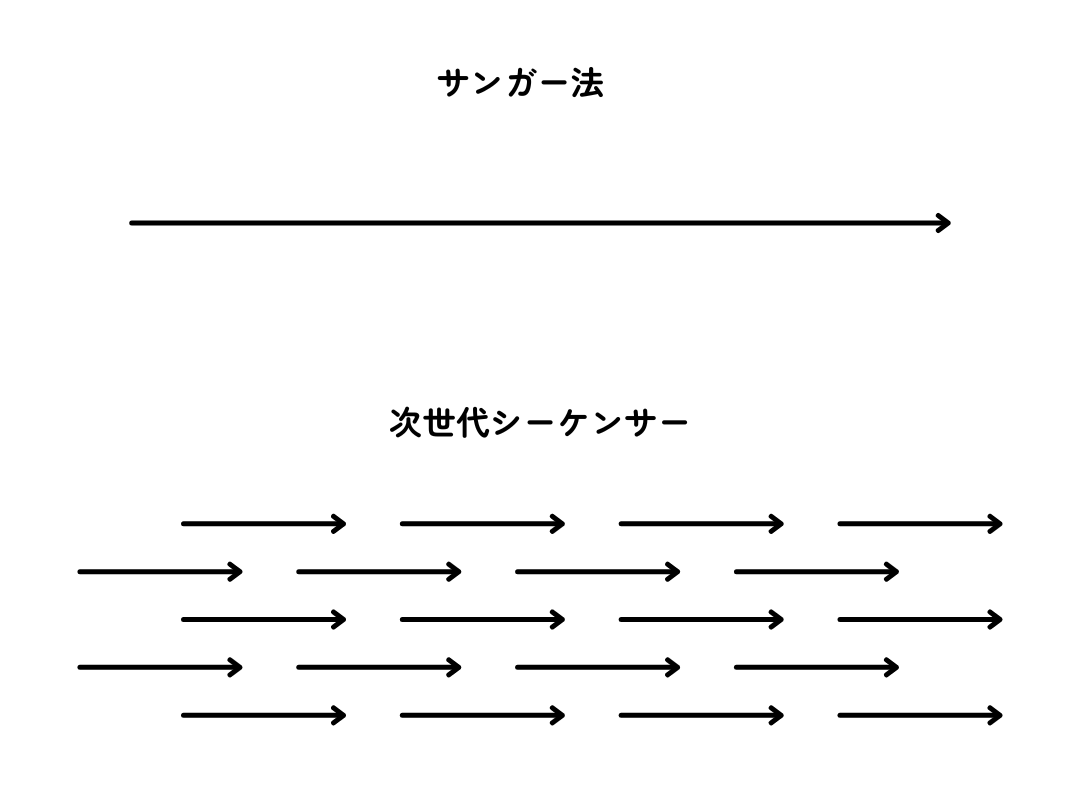
サンガー法では約1000塩基を解読でき、現在のサンガー法を使うシーケンサーは最大で16本のDNA、つまり1万6000塩基を同時に調べることができます。1回の操作で1〜2時間かかるため、1時間あたり1万塩基を調べるのが限界でしょう。
その後、2006年に登場したのが次世代シーケンサーです。次世代シーケンサーの詳しい原理や手順は後ほど解説しますが、サンガー法と次世代シーケンサーの最大の違いは処理能力(スループット)です。
次世代シーケンサーが解読できるのは150塩基程度と、サンガー法の約10分の1ですが、この短い配列を数百万から数億本集めて同時に解読します。1回の操作に1〜2日かかりますが、150塩基を5億本集めて24時間かけて解読すると仮定すると、1時間あたり約30億塩基を調べることができます。これはサンガー法の10万倍以上の処理能力です。
ちなみに、ヒトゲノムは30億塩基対からなります。サンガー法をベースにした方法で初めてヒトゲノムを解読した1990年代では、世界中の研究機関が協力して13年もかかりましたが、最新の次世代シーケンサーならたった1台で1日もあれば完了します。
上のイラストは、サンガー法と次世代シーケンサーの解読方法の比較です。サンガー法では合成したDNAの塩基配列を1本ずつ読みますが、次世代シーケンサーでは最大で数億本のDNAを同時に読むため、圧倒的に時間とコストを抑えることができます。
次世代シーケンサーでできること
次世代シーケンサーは多くの場面で使われています。代表例はヒトゲノム解析です。国内外で数万人から100万人規模で人々のゲノムを解析して、血液検査や病歴などの情報と合わせて疾患や体質などの個人差と遺伝情報の関連を明らかにしようとしています。日本では、東北メディカル・メガバンク機構(ToMMo)が日本人の一般住民集団を対象とした大規模なゲノム解析を行っており、5万4000人分の解析データをもとにして日本人のゲノム情報を公開しています。イギリスでは、約50万人のゲノムが解読されています。
また、医療現場でも次世代シーケンサーは使われています。最近のがん治療では、がんの進行に関わる特定のタンパク質にのみ作用するように設計された治療薬(分子標的薬)が使用されるようになってきています。この特定のタンパク質は、がん細胞で変異している遺伝子からつくられるのですが、どの遺伝子が変異しているのかを明らかにする際、がんに関係する遺伝子を一つずつ調べるのでは膨大な時間がかかってしまいます。そこで、次世代シーケンサーを使用すれば、一気に網羅的に遺伝子を調べることができるのです。
次世代シーケンサーの解読対象はヒトだけではなく、他の生物種のゲノムも解読できます。腸内細菌の研究が大きく進んでいますが、その一端を担っているのも次世代シーケンサーです。次世代シーケンサーは、異なる生物種が混ざったサンプルでも丸ごと解析して、どの生物種がどれくらいいるか解析できます。腸内細菌は、ヒトの腸内に何百種類といますが、その種類を簡単に分類できるのも次世代シーケンサーならではです。
次世代シーケンサーを使う手順
では、次世代シーケンサーを使った塩基配列決定の手順を解説します。なお、次世代シーケンサーは複数のメーカーから販売されており、それぞれ異なる原理や特徴があります。ここでは、多くの次世代シーケンサーを製造、販売している米国イルミナ社が採用している手順と原理を紹介します。次世代シーケンサーの手順には、ライブラリ調製、クラスター形成、シーケンシング、データ解析の4つのステップがあります。
【ステップ1】 ライブラリ調製
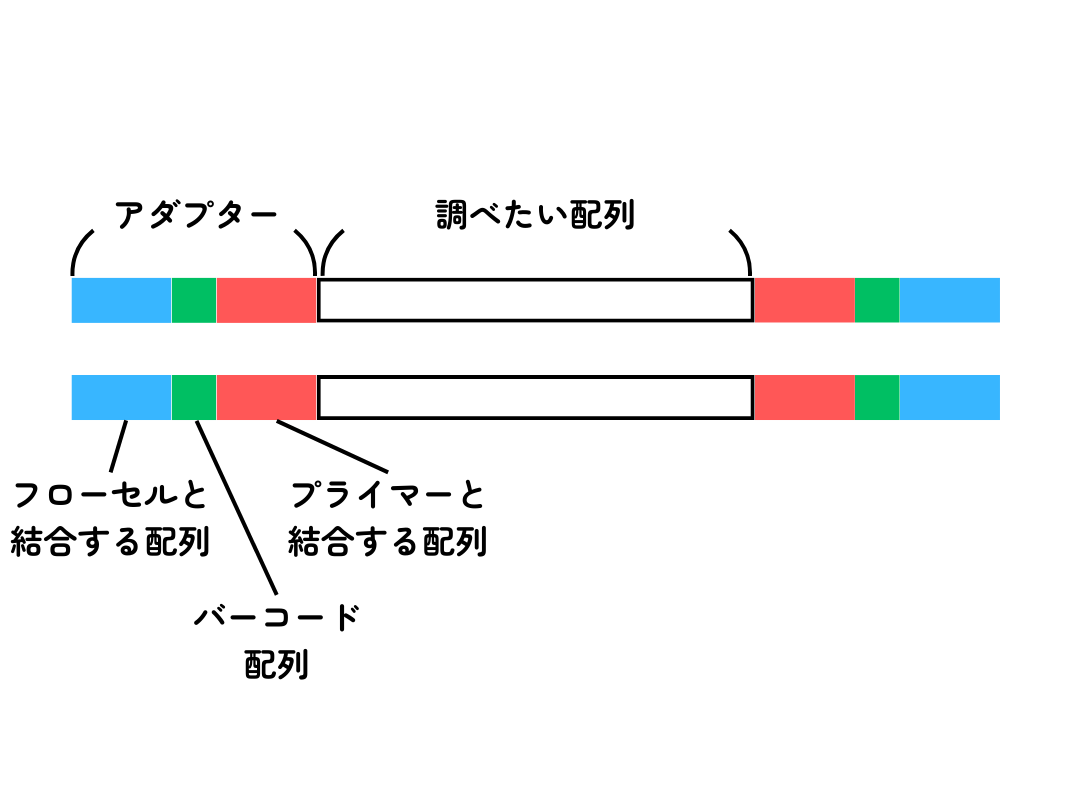
ライブラリ調製では、ゲノムDNAを断片化して、その両端にアダプターという配列をつなげます。このアダプターには、次のステップで使うフローセルと結合する配列、サンプル区別のためのバーコードとなる配列、プライマーというものが結合してDNA複製の開始点となる配列があります。
※ライブラリとは:同程度の長さのDNA断片の集まりで、5’末端と3’末端にそれぞれ配列既知のアダプターが付加された状態を指す。
※フローセルとは:ガラス基板上でライブラリのクラスターを形成するための土台
【ステップ2】クラスター形成
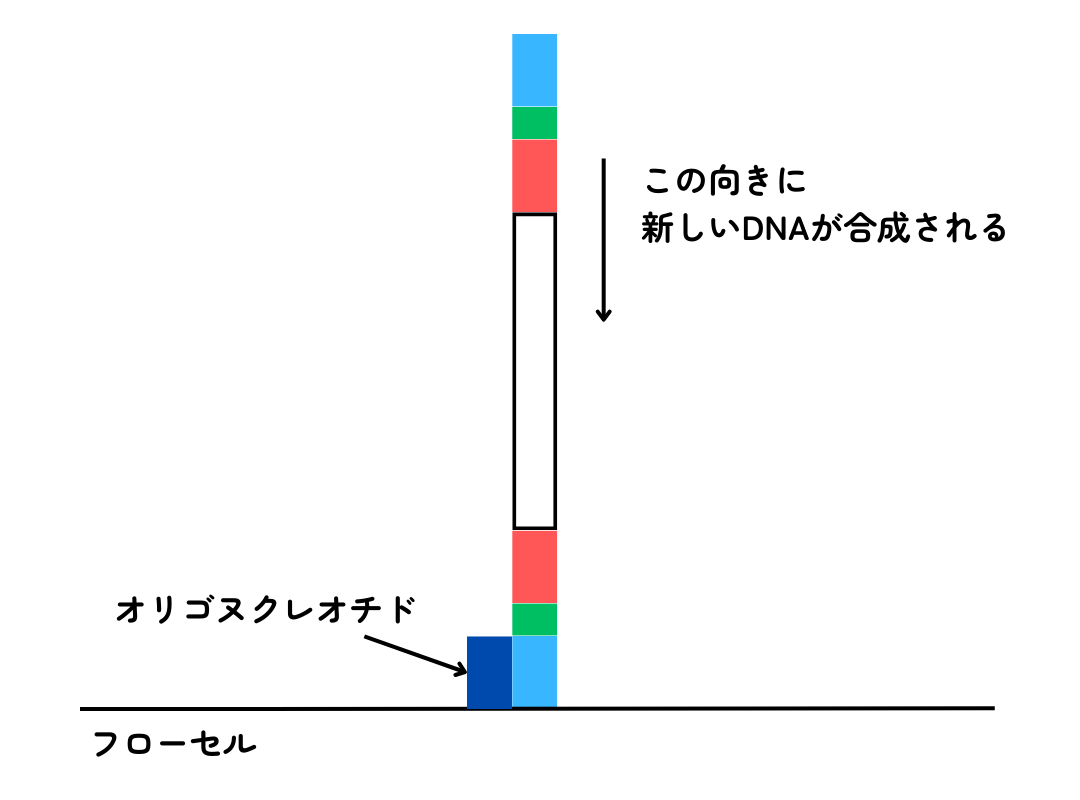
ステップ1のDNA断片を、フローセルという板に流し込みます。フローセルには、アダプター配列と結合できるオリゴヌクレオチド(短い1本鎖DNA)が多数埋め込まれており、そこにDNA断片が結合します。
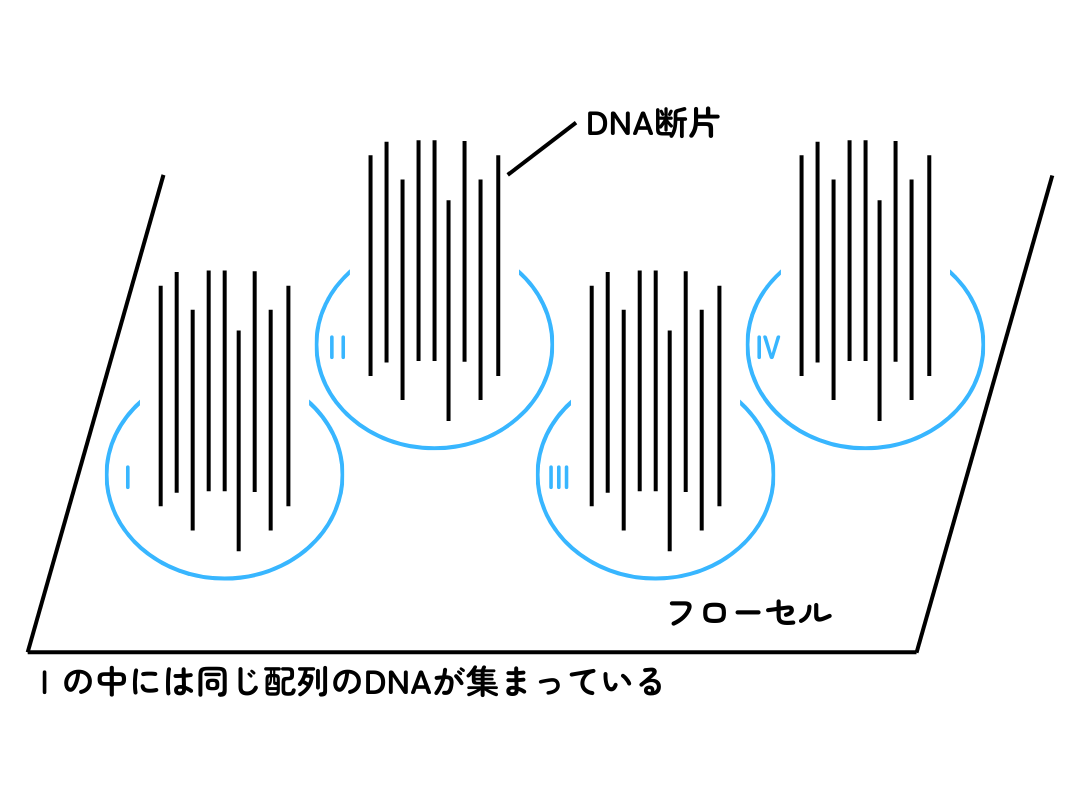
米国イルミナ社の次世代シーケンサーでは、ここでブリッジPCRという反応を使ってDNA断片を増幅させます。すると、同じ配列のDNA断片が1か所に集まった状態になります。この集まりをクラスターといいます。クラスターには、1個のDNA断片から増幅されて約1000個のDNA断片が集まっています。
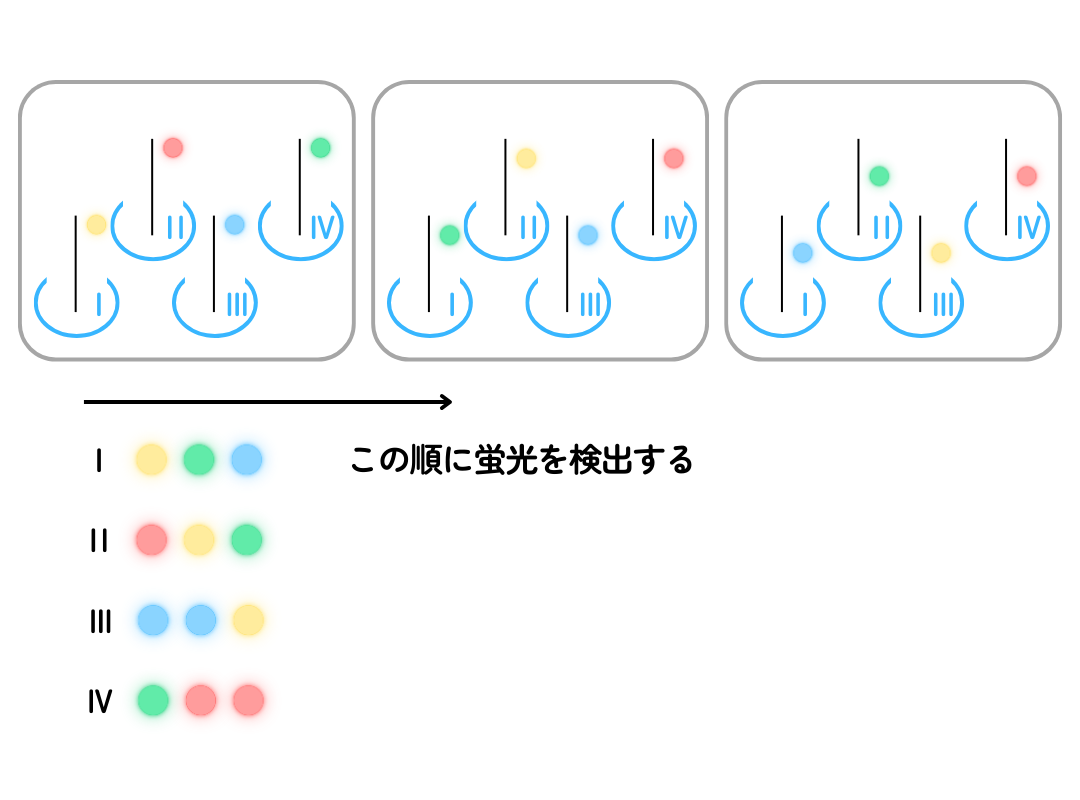
I、II、III、IVがそれぞれ別のクラスターになり、クラスターの中にあるDNAは同じ塩基配列をもちます。
【ステップ3】シーケンシング
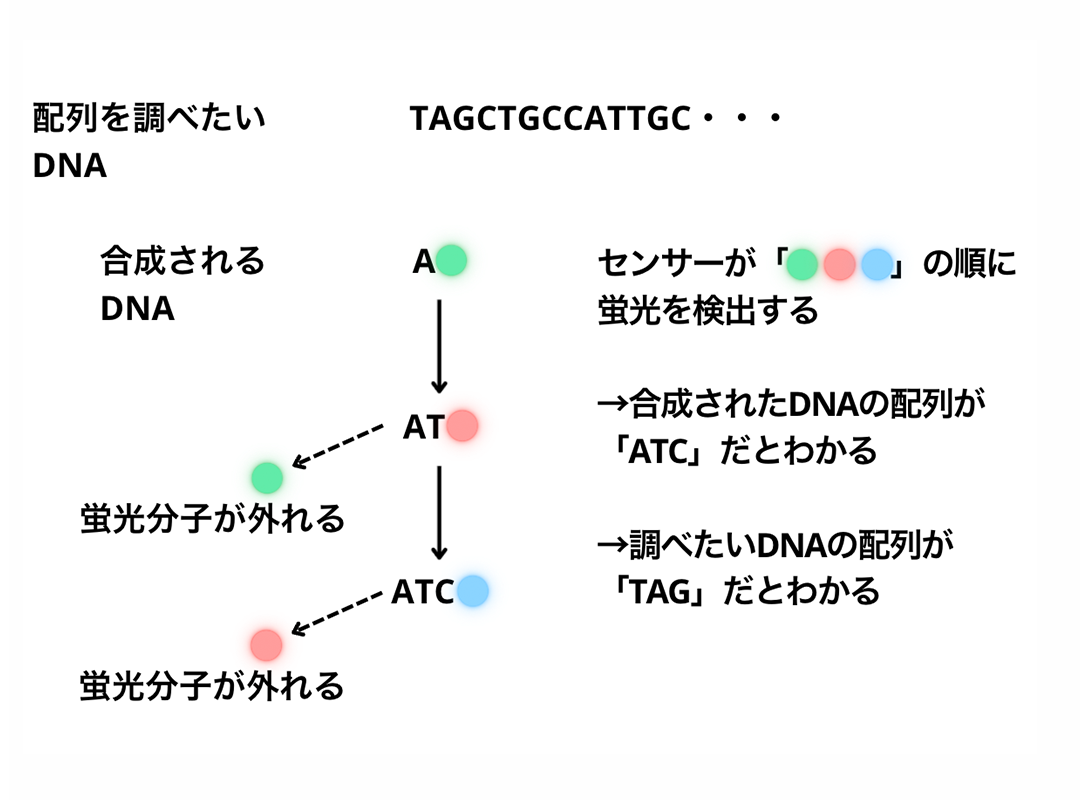
シーケンシングでは、DNA断片の配列をもとにして新しい配列をつくっていきます。このとき、4種類の塩基に対応して、異なる色を発する蛍光分子が結合したdNTP(デオキシリボヌクレオチド三リン酸)を使います。例えば、調べたいDNA断片の最初の塩基がTだった場合、相補的に結合するのはAになります。このとき、Aには黄色の蛍光分子が結合しています。次世代シーケンサーでは、1塩基が結合するごとに蛍光の色をセンサーで検出します。検出したら蛍光分子は外れ、次の塩基と蛍光分子がつながります。そして蛍光の色を検出する……を繰り返します。ステップ2でDNAを増幅させてクラスターを形成したのは、DNAの量を1000倍にして蛍光の明るさも1000倍にすることで蛍光を検出できるようにするためです。
次世代シーケンサーでは、DNAを合成しながら蛍光によって配列を調べます。クラスターにあるDNA断片の長さは150塩基程度ですが、1個のフローセルにつきクラスターは数百万から数億個もあります。そのため、一度に数億塩基から数百億塩基を調べることができ、30億塩基対からなるヒトゲノムを一度で全部解読できるのです。
フローセルを真上から見ると、クラスターごとに蛍光を発しているように見えます。白く囲ったところを拡大したものは次の図です。
例えばクラスターIでは、順に黄色、緑、青の順に蛍光を検出します。それぞれの蛍光色が4種類の塩基に対応しているため、塩基配列を知ることができます。
【ステップ4】 データ解析
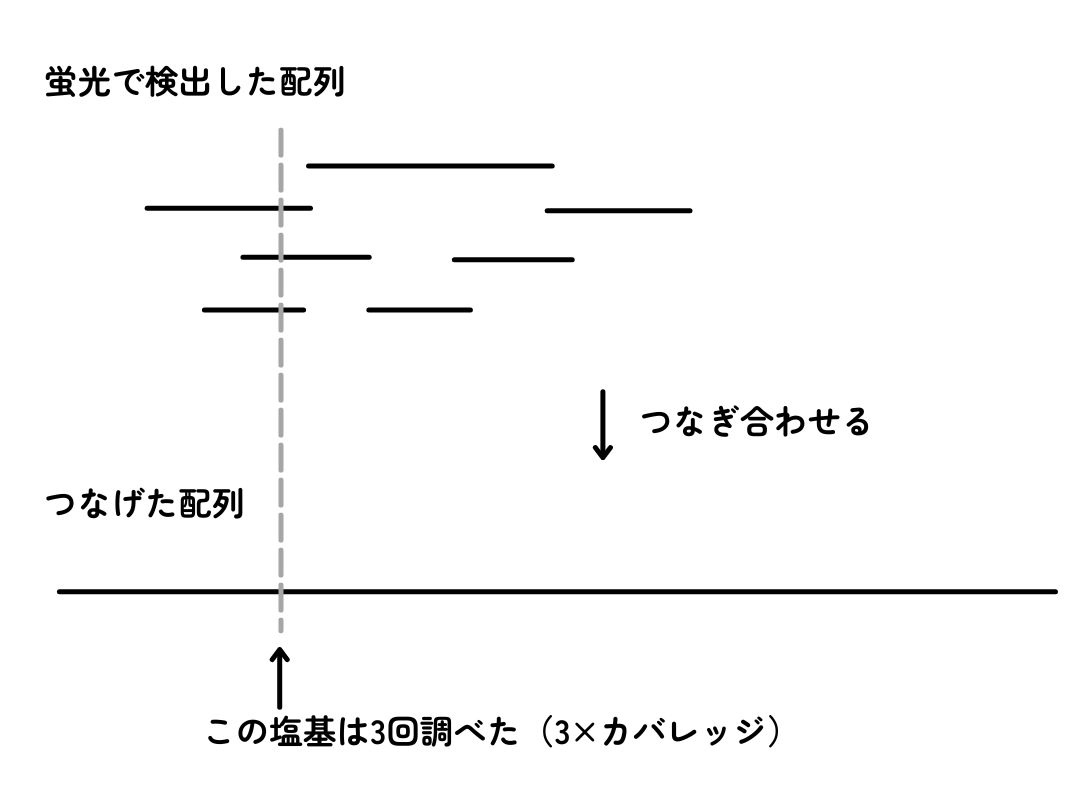
シーケンシングしたものは、ステップ1でゲノムDNAを断片化したものなので、最後につなぎ合わせる必要があります。重複する配列を手がかりに、コンピュータを用いて1つの配列として表示します。
なお、DNA複製ではどうしてもコピーミス(エラー)が発生します。そのため、DNA断片をいくつも重ねてエラーを最小化します。DNA断片をつなげ合わせ、ある塩基を決定する際、何本のDNA断片を使っているかの数を深度またはカバレッジといいます。例えば30×カバレッジで全ゲノムをシーケンスしたときには、平均して各塩基を30回シーケンスしたことを意味します。全ゲノムシーケンスでは30×から50×が求められます。
まとめ
次世代シーケンサーの登場によってDNAシーケンシングのスピードアップとコスト削減が劇的に進み、一度に大量にゲノム解析を行えるようになりました。その特徴を生かして、腸内細菌叢を丸ごと解析するなど、新しい研究手法も生まれています。
ところで、次世代シーケンサーがDNAシーケンサーの終着点ではありません。次世代シーケンサーとはまったく異なる原理で稼働する、いわば第3世代、第4世代のDNAシーケンサーも登場しています。例えば、米パシフィック・バイオサイエンシズ・オブ・カリフォルニア社のシーケンサーは、DNAを断片化せずに数万塩基をそのまま解読できるのが特徴です。これとはまた違う原理を採用するのが英オックスフォード・ナノポア社です。USBで稼働するポータブルサイズのシーケンサーもあり、研究室から離れたフィールドワークでのDNA解析も可能にしています。
今後も次世代シーケンサーを含めたDNAシーケンサーの技術発展により、生命科学や医学がさらに発展すると期待されます。
記事執筆:島田祥輔(名古屋大学大学院理学研究科生命理学専攻修士課程修了 / サイエンスライター)
(上記すべて参照:2024-2-1)