化学実験、バイオ実験のノウハウなど、毎日の実験・分析に役立つ情報をお届け。

この記事は、理系研究職の方のキャリア支援を行うパーソルテンプスタッフ研究開発事業本部(Chall-edge/チャレッジ)がお届けする、実験ノウハウシリーズです。
PCR生成物の定量的な解析法(定量的PCR; quantitative PCR/qPCR)
遺伝子の発現解析といえば、従来はノーザンブロット法が主流でした。その一方、ノーザンブロット法は感度が低く、一過性に発現する遺伝子など、低コピー数のmRNAを解析対象とする場合には、常に大きな困難を伴いました。やがて、感度と特異性がともに高いPCRをもとにした遺伝子発現の解析法(RT-PCR法)[前項参照]が開発され、ノーザンブロットは主役の座を奪われていきました。
しかしながら、エンドポイントPCRと呼ばれる通常のPCRでは、観察点(サイクル数)によっては、PCR生成物の量が必ずしも初期鋳型DNA量を反映しないため[PCRの項 参照]、初期鋳型量(mRNA発現量)の定量的な推定にはPCRの反応動態(カイネティクス)の理解が必須となります。ここでは、PCR生成物の定量的な解析法(定量的PCR; quantitative PCR/qPCR)について学んでいきましょう。
1. PCRの力イネティクスをもとにした指数関数的増幅期でのPCR生成物の定量
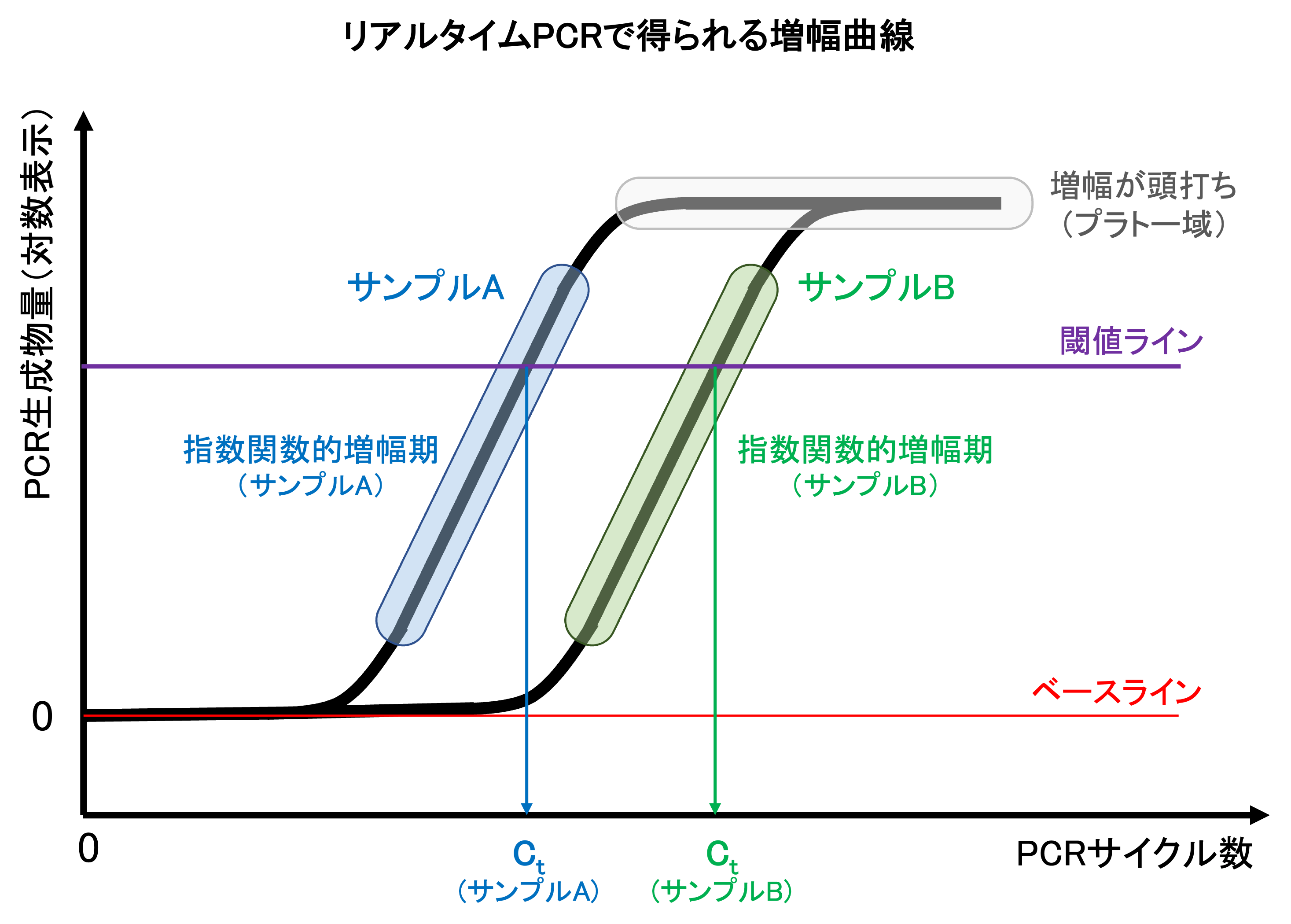
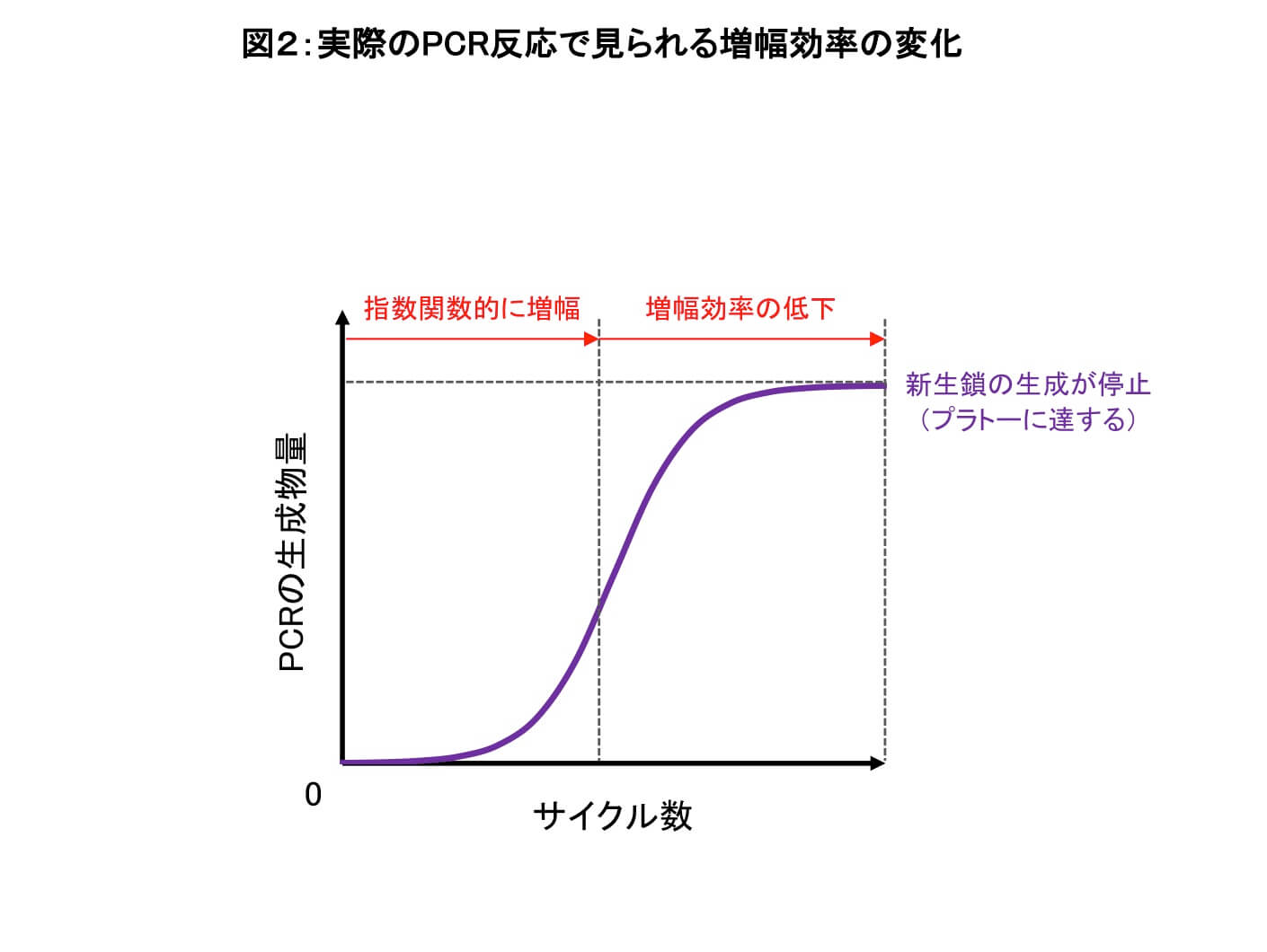
PCRのサイクル数を横軸に、生成物量の対数を縦軸にプロットした増幅曲線では、PCR生成物は反応初期にはほぼ指数関数的に増幅するが、ある一定以上のPCRサイクル数ではDNAの増幅が頭打ち(プラトー)になる。このPCR増幅反応がプラトーに達しているサイクル数は、PCRの定量解析に不適となります。
片対数のPCR増幅曲線で、生成物量がサイクル数に対して直線性を示す範囲は、PCR反応がプラトーに達する前の指数関数的増幅期であり、nサイクル目の生成物の量(y)は以下の理論式 [3] で算出することができる[PCRの項 参照] 。
y = I X En (1 ≤ E ≤ 2 , I = 初期鋳型量, E = PCRの増幅効率) ・・・・・・・・・・・・ [3]
以上より、PCRの定量的解析は、指数関数的増幅期で生成物量を測定・比較することで初めて可能となります。しかしながら、実際のPCRでは、目的DNAの量(初期鋳型量)は未知であり、定量的な解析ができるPCRサイクル数の範囲を事前に知ることは不可能です。 指数関数的増幅期を知るためには、複数のエンドポイント、すなわち異なるPCRサイクル数で生成物量を測定し、PCR反応の力イネティクスを明らかにします。
まとめると、複数のサンプル間における初期鋳型DNAの定量比較は以下のように行うことになります。
- 測定する全てのサンプルについて、増幅曲線の片対数グラフが直線となるPCRサイクル数(指数関数的増幅期)を求める。
- 全てのサンプルについて、指数関数的増幅期内で、PCR生成物量とそれに対応するサイクル数を求め、これらをもとに初期鋳型量の比を算出する。
2. リアルタイムPCRによる相対定量と絶対定量
- 一般的な定量的PCRでは、反応系に存在する目的DNAの相対量を明らかにすること(相対定量)はできるが、その絶対量を直接測定すること(絶対定量)はできない。
→サンプルAとサンプルBの初期鋳型量に何倍の差があるかを求めることはできるが、サンプルAに何pgの目的DNAが存在するかは直接的には測定できない。
→絶対量を求めるためには、絶対量が既知のDNA標準液を用いた定量的PCRを事前に行い、得られた検量線をもとに、間接的に目的DNAの絶対量を算出する(絶対定量)。 - 定量結果から意味のある結論を得るためには、目的DNAの測定と同時に、反応系に加えたサンプルの量を同時に測定して、これをもとに相対定量を行う必要がある。
→サンプル量を反映するような内部標準(インターナル・コントロール)DNAの測定値を用いて、目的DNAの測定値を補正することで、目的DNAの定量結果を正当に評価できるようになる。
<例> 2つのサンプルAとBにおける遺伝子Xの発現量を比較する場合
遺伝子Xの定量結果が、A : B = XA : XB、
内部標準の遺伝子Nの定量結果が、A : B= NA: NBだったとき、
内部標準Nで補正した遺伝子Xの発現量は、 A : B = XA/NA : XB/NB となる。
<内部標準となるDNAの例>
- ハウスキーピング遺伝子
組織間や刺激の有無で発現量が変化しない遺伝子(グリセロアルデヒドリン酸脱水素酵素(GAPDH)、βアクチン、γアクチン、リボソーム26Sなど)。対象の内部標準遺伝子が、サンプル間で同等に発現していることを事前に確認する。 - 実験者が反応系に添加した核酸
内部標準となる内在性の遺伝子が得られない場合は、PCR反応系に標準となる核酸を添加します。全サンプルに一定量の標準DNA/RNAをあらかじめ加えてからPCRサンプルを調整し、目的DNAおよび標準DNAの定量的PCRを行います。
3. PCR生成物の定量解析に用いる蛍光レポーターの選択
- DNAインターカレーターSYBR Green Iを用いた検出
PCR生成物のレポーターとして、DNAにインターカレートする蛍光色素、SYBR Green Iが用いられている。SYBR Green Iは、非特異的なバックグラウンド蛍光が低く、エチジウムブロマイド(EtBr)と比較して変異原性が低く、感度が10倍程度高い特性をもつ。約260 nmの紫外線もしくは約470 nmの可視光で励起されて緑色蛍光を発します。
- TaqManプローブを用いた検出
PCR増幅断片の一部分と相補的な配列を持つオリゴヌクレオチドプライマーに、2種の蛍光分子(レポーターとクエンチャー)を付加したTaqManプローブを事前に作製する。このプローブをPCR反応系に導入すると、PCR生成物の量に比例した蛍光強度がプローブを取り込んだPCR生成物から得られる。PCR生成物を染色する方法に比べて、バックグラウンドが低く、検出感度が高い特長をもつ一方、配列の異なるPCR断片ごとに異なるプローブを設計する必要がある。
次項では、これらの注意点に気をつけて、実際に定量的PCRを行ってみましょう。
*監修
パーソルテンプスタッフ株式会社
研究開発事業本部(Chall-edge/チャレッジ)
研修講師(理学博士)
この記事は、理系研究職の方のキャリア支援を行うパーソルテンプスタッフ研究開発事業本部(Chall-edge/チャレッジ)がお届けする、実験ノウハウシリーズです。
過去の記事一覧:実験レシピシリーズ
関連記事Recommend
-

細胞の免疫蛍光染色 実践編(2/2)
リケラボ実験レシピシリーズ
-

細胞の免疫蛍光染色 基本編(1/2)
リケラボ実験レシピシリーズ
-

細胞内でRNA干渉を人為的に誘導するために RNA干渉による遺伝子発現の調節(2/2)
リケラボ実験レシピシリーズ
-

RNA干渉(RNA interfering, RNAi)とは? RNA干渉による遺伝子発現の調節(1/2)
リケラボ実験レシピシリーズ
-

免疫沈降法(Immunoprecipitation)による目的タンパク質の濃縮 基本から応用まで
リケラボ実験レシピシリーズ
-

ウェスタンブロットによるタンパク質の発現解析(3/3)ウェスタンブロットによるタンパク質の検出
リケラボ実験レシピシリーズ
-

ウェスタンブロットによるタンパク質の発現解析(2/3)培養細胞抽出液のSDSポリアクリルアミド電気泳動
リケラボ実験レシピシリーズ
-

ウェスタンブロットによるタンパク質の発現解析(1/3)タンパク質実験をはじめるための基礎知識
リケラボ実験レシピシリーズ
-

リポフェクション法による遺伝子導入のプロトコル
リケラボ実験レシピシリーズ トランスフェクション(2/2)
-

遺伝子導入技術 トランスフェクションの基礎知識
リケラボ実験レシピシリーズ トランスフェクション(1/2)